סליל כפול של חומצות גרעין
בביולוגיה מולקולרית, המונח "סליל כפול"[1] מתייחס למבנה שנוצר על ידי מולקולות דו-גדיליות של חומצות גרעין כגון דנ"א. המבנה הסלילי הכפול של קומפלקס חומצות הגרעין נובע כתוצאה מהמבנה השניוני שלו, והוא מרכיב יסודי בקביעת המבנה השלישוני שלו. המונח נכנס לתרבות הפופולרית עם פרסום הספר "הסליל הכפול" מאת ג'יימס ווטסון (1968) העוסק בגילוי מבנה הדנ"א.
הסליל הכפול של הדנ"א הוא פולימר חומצות גרעין המורכב מנוקלאוטידים, שיוצרים יחד זוגות קבועים.[2] ב-B-DNA, קונפורמציית הסליל הכפול הנפוצה ביותר בטבע, הסליל הכפול הוא סליל ימני עם כ-10 עד 10.5 זוגות בסיסים לכל פיתול.[3] מבנה הסליל הכפול של הדנ"א מכיל תעלה גדולה (Major Groove) ותעלה קטנה (Minor Groove). בגאומטריית B-DNA התעלה הגדולה רחבה יותר מאשר התעלה הקטנה.[2] בשל הבדל זה, חלבונים רבים שנקשרים ל-B-DNA עושים זאת דרך התעלה הגדולה.[4]
היסטוריה
מודל הסליל הכפול של מבנה הדנ"א פורסם לראשונה בכתב העת Nature על ידי ג'יימס ווטסון ופרנסיס קריק ב-1953.[5][6] הוא מבוסס על צילום הרנטגן החשוב של הדנ"א שכותרתו "תצלום 51 ", שצילמה רוזלינד פרנקלין ב 1952,[7] ומאוחר יותר על תצלום בהיר יותר של הדנ"א שצילמה יחד עם ריימונד גוסלינג,[8][9] מוריס וילקינס, אלכסנדר סטוקס והרברט וילסון,[10] ועל מידע כימי וביוכימי הנוגע לזיווג הבסיסים שהוסיף ארווין צ'רגף.[11][12][13][14][15][16] לפני כן מודל הדנ"א הרווח הכיל שלושה גדילים.[17]
ההבנה שהדנ"א בנוי מסליל כפול, ועימה הבהרת מנגנון זיווג הבסיסים בו מידע גנטי מאוחסן ומועתק באורגניזמים חיים, נחשבת לאחת התגליות המדעיות החשובות ביותר של המאה ה-20. בשנת 1962 ווילקינס, ווטסון וקריק קיבלו על תגלית זו שליש פרס נובל כל אחד בפיזיולוגיה או רפואה על תרומתם לגילוי.[18] פרנקלין, אשר תצלום הרנטגן פורץ הדרך שהפיקה סיפק נתונים חשובים לגיבוש מבנה הדנ"א, נפטרה בשנת 1958 ולכן לא הייתה זכאית להיות מועמדת לפרס נובל.
היברידיזציה של חומצות גרעין
היברידיזציה (הכלאה) היא תהליך בו שני גדילים משלימים נקשרים זה לזה ליצירת סליל כפול. ניתן להביא להיברידיזציה של דנ"א משני מקורות שונים על ידי דנטורציה (התכה) של דנ"א דו-גדילי משני המקורות והשרייתם יחד בתנאים המתאימים. שיטה זו משמשת להשוואה בין שתי דגימות דנ"א או דגימת דנ"א ורנ"א, למשל משני אורגניזמים שונים, או איתור של מקטע דנ"א ספציפי. ניתן להביא לדנטורציה של חומצות גרעין על ידי חימום או חשיפה לבסיסים חזקים, בהם נשברים קשרי המימן בין בסיסים מזווגים וקשרי המערום בין בסיסים סמוכים.[19] באופן זה שני הגדילים של הסליל הכפול נפרדים, אך הקשרים הקוולנטים שבין הבסיסים באותו הגדיל אינם מופרעים ושלמות הגדילים לא נפגעת.
דנטורציה של חומצות גרעין באמצעות חימום עדין, כמו זו המתבצעת בתגובת שרשרת של פולימראז (PCR), אידיאלית כאשר מולקולות חומצות הגרעין מורכבות מפחות מ-10,000 זוגות בסיסים. השזירה של גדילי הדנ"א מקשה על הפרדת מקטעי דנ"א ארוכים מכך. התא מתגבר על בעיה זו באמצעות אנזימים מקבוצת הליקאז, המאפשרים עבודה מקבילית עם האנזים טופואיזומראז, החותך את הגב הפוספו-זרחני של אחד מהגדילים. הליקאז פורמים את הגדילים ומאפשרים את השלמת גדילי הדנ"א, למשל באמצעות דנ"א פולימראז.
גאומטריה של זוג בסיסים
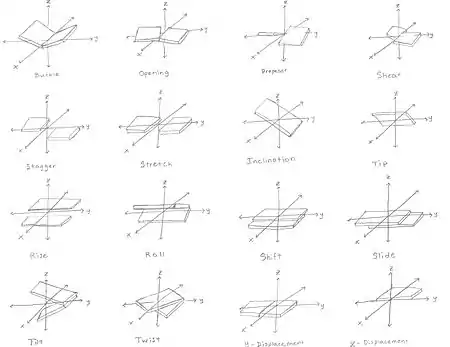
את הגאומטריה של הבסיס, או של זוג בסיסים, ניתן לאפיין באמצעות 6 קואורדינטות: העתקה, החלקה, התרוממות, הטיה, גלגול ופיתול. ערכים אלו מגדירים במדויק את המיקום והאוריינטציה במרחב של כל בסיס או זוג בסיסים במולקולה של חומצת גרעין ביחס לקודמו לאורך ציר הסליל. במונחים של דנ"א או רנ"א, כאשר המבנה הרצוי נפגע, ניתן לתאר את ההפרעה היטב באמצעות שימוש במונחים אלה.
עבור כל זוג בסיסים, המחושב ביחס לקודמו, קיימות הגאומטריות הבאות:[20][21][22]
- גזירה (Shear)
- מתיחה (Stretch)
- תנודה (Stagger)
- רכיסה (Buckle)
- מדחף (Propeller): סיבוב של בסיס אחד ביחס לשני באותו זוג בסיסים.
- פתיחה (Opening)
- העתקה (Shift): תזוזה לאורך ציר המישור של זוג בסיסים בניצב לראשון, המופנה מהתעלה הקטנה לתעלה הגדולה.
- החלקה (Slide): תזוזה לאורך ציר המישור של זוג בסיסים המופנית מגדיל אחד לשני.
- התרוממות (Rise): תזוזה לאורך ציר הסליל.
- הטייה (Tilt): סיבוב סביב ציר ההעתקה.
- גלגול (Roll): סיבוב סביב ציר ההחלקה.
- פיתול (Twist): סיבוב סביב ציר ההתרוממות.
- עקירת-x (באנגלית x-displacement)
- עקירת-y (באנגלית y-displacement)
- הרכנה (Inclination)
- שיפוע (Tip)
- גובה (Pitch): מספר זוגות הבסיסים לכל סיבוב שלם של הסליל.
ההתרוממות והפיתול קובעות את כיוון הסיבוב (ימני/שמאלי) ואת גובהו של הסליל והן היחידות מבין הקואורדינטות שלא יכולות להיות שוות לאפס. ערכי ההעתקה וההחלקה בצורה B הם לרוב קטנים, אך משמעותיים יותר בצורות A ו-Z. הגלגול וההטייה לרוב קטנים ומעידים על זוגות בסיסים פחות מקבילים.
שימו לב שבספרות המדעית, לעיתים קרובות נעשה שימוש במונח "הטייה" (tilt) בהתייחס להסט הציר הבין-גדילי הראשון מהניצב לציר הסליל. מונח זה מקביל להחלקה בין זוגות בסיסים רציפים, ובקורדינטות מבוססות סליל המינוח התקין הוא "הרכנה" (Inclination).
הגאומטריה של הסליל
בטבע מוכרות שלוש קונפורמציות של דנ"א: A-DNA ,B-DNA ו-Z-DNA. צורה B של דנ"א היא זו שתוארה על ידי ג'יימס ווטסון ופרנסיס קריק והיא נחשבת לנפוצה ביותר בטבע והיציבה ביותר בתנאים פיזיולוגיים.[23] קוטר צורה B הוא 23.7 אנגסטרום (Å) וגובהה 3.4 אנגסטרום לכל זוג בסיסים. הסליל הכפול משלים סיבוב אחד סביב צירו כל 10.4-10.5 זוגות בסיסים. תדירות פיתול זו תלויה במידה רבה באינטראקציות המערום שכל בסיס מפעיל על שכניו בשרשרת. התצורה המוחלטת של הבסיסים קובעת את כיוון פיתול הסליל עבור כל קונפורמציה.
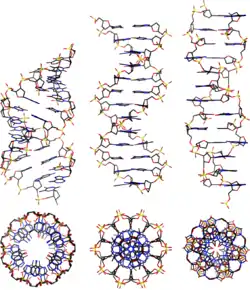
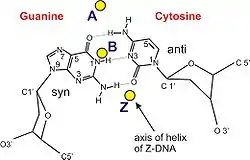
צורת A-DNA וצורת Z-DNA נבדלות באופן משמעותי בגאומטריה שלהן ובממדיהן מצורה B, אך כל שלוש הקונפורמציות יוצרות מבנה של סליל כפול. צורה A יציבה יותר בסביבות דלות במים, ובמשך זמן רב סברו כי צורה זו מופיעה רק בדגימות מעבדה מיובשות של דנ"א, כגון אלה המשמשות ניסויים קריסטלוגרפיים, וכן בהיברידיזציה של גדילי דנ"א ורנ"א. בהמשך התגלה כי התייבשות דנ"א מתרחשת גם in vivo, וצורה A מוכרת כיום כבעלת תפקודים ביולוגיים. צורה A וצורה B הן שתיהן בעלות כיוון סיבוב ימני. קטעי דנ"א שעברו מתילציה בתאים למטרות רגולטוריות עשויים לאמץ צורת Z, בה הגדילים הופכים את כיוון סיבוב הסליל לשמאל, ויש עדויות לקיומם של קומפלקסים של דנ"א-חלבון היוצרים מבנים בצורת Z.
קיימות קונפורמציות אפשריות נוספות; C-DNA ,E-DNA,[24] L-DNA (צורה אננטיומרית של D-DNA),[25] P-DNA,[26] S-DNA ועוד תוארו עד כה.[27] למעשה, רק האותיות F/V/U/Q/Y זמינות לתיאור קונפורמציות דנ"א חדשות שעשויות להתגלות בעתיד.[28][29] עם זאת, כלל הקונפורמציות מלבד B ,A ו-Z נוצרו באופן סינתטי ולא נצפו במערכות ביולוגיות בטבע. קיימים גם מודלים של דנ"א תלת-גדילי וצורות Guadruplex כגון G-quadruplex ו-i-motif .
| מאפיין הגאומטריה | A-DNA | B-DNA | Z-DNA |
|---|---|---|---|
| כיוון הסיבוב | ימני | ימני | שמאלי |
| יחידה חוזרת | 1 bp | 1 bp | 2 bp |
| סיבוב / bp | 32.7° | 34.3° | 60°/2 |
| מספר זוגות בסיסים לסיבוב | 11 bp | 10.5 bp | 12 bp |
| ההטיה ביחס לציר הסליל | +19° | -1.2° | -9° |
| גובה כל זוג בסיסים | 2.3 Å
(0.23 ננומטר) |
3.32 Å
(0.332 ננומטר) |
3.8 Å
(0.38 ננומטר) |
| קונפורמציית הקשר הגליקוזילי | אנטי | אנטי | פירמינים: אנטי, פורינים: סין. |
| קונפורמציית קיפול טבעת הסוכר | C3'-אנדו | C2'-אנדו | פירמינים: C2'-אנדו,
פורינים: C3'-אקסו. |
| קוטר | 23 Å
(2.3 ננומטר) |
20 Å
(2.0 ננומטר) |
18 Å
(1.8 ננומטר) |
תעלות
גדילי סליל תאומים יוצרים את השלד של הדנ"א. ניתן למצוא סליל כפול נוסף על ידי מעקב אחר הרווחים, או התעלות, בין הגדילים. חללים אלה סמוכים לזוגות הבסיסים ויכולים לספק אתר קישור. בשל כך שהגדילים אינם מנוגדים לחלוטין זה לזה, התעלות אינן בגודל שווה: רוחב התעלה הגדולה (הגדולה) הוא 22 אנגסטרום ורוחב התעלה הקטנה (הקטן) 12 אנגסטרום.[33] צרותה של התעלה הקטנה מביאה לכך שקצוות הבסיסים נגישים יותר בתעלה הגדולה. כתוצאה מכך, חלבונים כדוגמת גורמי שעתוק שנקשרים לרצפים ספציפיים בדנ"א דו-גדילי לרוב יוצרים קשרים בצידי הבסיסים שנחשפו בתעלה הגדולה.[4] מצב זה משתנה בהתאם לקונפורמציות החריגות של הדנ"א בתוך התא, אך בכל מקרה התעלות העיקריות והמשניות תמיד יקראו כך שישקפו את הבדלי הרוחב שהיו נמדדים לו הדנ"א היה נשזר בצורת B הסטנדרטית.
צורות לא סליליות כפולות
בסוף שנות ה-70', מודלים לא סליליים אלטרנטיביים של דנ"א נחשבו לזמן קצר כפתרון פוטנציאלי לבעיות בשכפול דנ"א בפלסמידים ובכרומטין. מודלים אלו נזנחו לטובת מודל הסליל הכפול בעקבות ההתקדמות הניסויית בקריסטלוגרפיה בקרני רנטגן של דופלקסים של דנ"א ולאחר מכן גילוי הנוקלאוזום והטופואיזומראז. כיום, המודלים הלא-סליליים אינם מקובלים על ידי הקהילה המדעית.[34][35]
חומצות גרעין חד-גדיליות (ssDNA) אינן מסתדרות במבנה סלילי, ומתוארות על ידי מודלים כגון ליפוף אקראי או שרשרת דמוית תולעת.
כיפוף
אורך התמדה, קשיחות צירית
| סדר פעולות | אורך התמדה / זוגות בסיסים |
|---|---|
| אקראי | 154 ± 10 |
| (CA) חוזר | 133 ± 10 |
| (CAG)חוזר | 124 ± 10 |
| (TATA) חוזר | 137 ± 10 |
הדנ"א הוא פולימר קשיח יחסית, הממודל לרוב כשרשרת דמוית תולעת (WLC). יש לו שלוש דרגות חופש עיקריות: כיפוף, פיתול ודחיסה, כל אחת מהן מגבילה בצורה אחרת את שימושי הדנ"א האפשריים בתוך התא. קשיחות סיבוב הפיתול (Twisting-Torsional Stiffness) של מולקולות דנ"א חשובות עבור המעגליות שלהן והתמצאות חלבונים קושרי דנ"א אחד ביחס לשני.
קשיחות ציר הכיפוף חשובה עבור שזירת הדנ"א, מעגליותו ואינטראקציות בין חלבונים. בהיעדר מתח גבוה, תוספות דחיסה אינן חשובות באופן יחסי.
דנ"א בתמיסה אינו מאמץ מבנה קשיח אלא עובר ללא הרף בין קונפורמציות בהשפעת רטט תרמי והתנגשויות עם מולקולות המים. מעברים אלו הופכים את המדידות הקלאסית של קשיחות לבלתי אפשריות ליישום. לפיכך, קשיחות הכיפוף של הדנ"א נמדדת על ידי אורך התמדה (Persistence Length), תכונה מכנית בסיסית באמצעותה ניתן לאמוד את קשיחות הפולימר.
ניתן למדוד ערך זה ישירות באמצעות מיקרוסקופ כוח אטומי להדמיה ישירה של מולקולות דנ"א באורכים שונים. בתמיסה מימית, אורך ההתמדה הממוצע הוא 46–50 ננומטר או 140–150 זוגות בסיסים (קוטר הדנ"א הוא 2 ננומטר), אם כי טווח השינויים רחב. הדנ"א היא מולקולה קשיחה למדי.
אורך ההתמדה של קטע הדנ"א תלוי במידה מסוימת בריצוף שלו, שביכולתו לחולל שינויים משמעותיים. השונות נגרמת בעיקר בשל אינטראקציות מערום בין הבסיסים והשיירים המשתרעים על פני התעלות הגדולה והקטנה.
מודלים עבור כיפוף דנ"א
| זוג הבסיסים | כוחות מערום ΔG/kcal mol -1 |
|---|---|
| T A | -0.19 |
| T G או C A | -0.55 |
| C G | -0.91 |
| A G או C T | -1.06 |
| A A או T T | -1.11 |
| A T | -1.34 |
| G A או T C | -1.43 |
| C C או G G | -1.44 |
| A C או G T | -1.81 |
| G C | -2.17 |
הגמישות האנטרופית של הדנ"א עולה בקנה אחד עם המודלים הסטנדרטיים לפיזיקה של פולימרים, כגון מודל שרשרת דמוי תולעת (WLC). תאימות עם מודל WLC ניכרת בכך שכיפוף הדנ"א מקיים את חוק הוק ביחידות מידה קטנות במיוחד (חלקי פיקוניוטון). עם זאת, עבור קטעי דנ"א שקצרים יותר מאורך ההתמדה, כוח הכיפוף הוא כמעט קבוע והתנהגותם חורגת מתחזיות מודל WLC.
תופעה זו גורמת למולקולות דנ"א קטנות להתעגל בקלות רבה ומעלה את ההסתברות למציאת מקטעי דנ"א מכופפים מאוד.
כיפוף מועדף
לעיתים קרובות, למולקולות הדנ"א יש כיוון כיפוף מועדף, כלומר הכיפוף הוא אנאיזוטרופי. זאת בשל התכונות של הבסיסים מהם מורכב קטע הדנ"א - לרצף אקראי לא יהיה כיוון כיפוף מועדף, כלומר הכיפוף יהיה איזוטרופי.
כיוון כיפוף דנ"א מועדף נקבע על ידי יציבות אינטראקציות המערום בין הבסיסים. כאשר בסיסים היוצרים אינטראקציות מערום לא יציבות נמצאים תמיד בצד אחד של הסליל הכפול, אזי הכיפוף המועדף של המולקולה יהיה לצד השני. ככל שזווית הכיפוף גדלה, השפעתם של ההפרעה הסטרית ושל חופש הסיבוב של השיירים בינם לבין עצמם עולה, במיוחד בתעלה הקטנה: שיירי A ו-T יעדיפו להימצא בתעלות הקטנות בחלק הפנימי של העיקולים. השפעות אלו בולטות במיוחד בחלבונים קושרי דנ"א בהם מושרה כיפוף הדוק, כגון בחלקיקי נוקלאוזום.
מולקולות דנ"א עם כיפוף מועדף משמעותי יכולות להיות מכופפות במיוחד. אלו נצפו לראשונה בדנ"א קינטופלאסטי (kDNA) של טריפנוסומטיד. רצפים אופייניים שגורמים לכך מכילים רצף של 4–6 שיירי T ו-A המופרדים במקטעים עשירים בשיירי G ו-C, ששומרים על שיירי ה-A וה-T בפאזה עם התעלה הקטנה בצד אחד של המולקולה. לדוגמה:
| ¦ | ¦ | ¦ | ¦ | ¦ | ¦ | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| G | A | T | T | C | C | C | A | A | A | A | A | T | G | T | C | A | A | A | A | A | A | T | A | G | G | C | A | A | A | A | A | A | T | G | C | C | A | A | A | A | A | A | T | C | C | C | A | A | A | C |
הכיפוף המשמעותי במבנה מושרה על ידי "פיתול מדחף" (Propeller Twist) של זוג בסיסים אחד ביחס לשני, המאפשר יצירה של קשרי מימן לא טריוויאליים בין בסיסים מזוגות שונים. כאשר הטמפרטורה עולה מספיק, הסליל הכפול עובר דנטורציה והכיפוף המשמעותי נהרס.
לכל דנ"א בעל כיפוף אנאיזוטרופי יש בממוצע אורך התמדה ארוך יותר וקשיחות צירית גבוהה יותר. אלו מביאים לנוקשות מוגברת שנדרשת על מנת למנוע כיפופים רנדומאליים שיגרמו למולקולה להתנהג בצורה איזוטרופית.
מחזוריות
מעגלי דנ"א תלוית הן בקשיחות ציר הכיפוף והן בקשיחות הסיבובית (Torsional) של המולקולה. בשביל שמולקולת דנ"א תהיה מעגלית עליה להיות ארוכה מספיק בשביל להתעקם בקלות למעגל שלם ולהכיל מספר בסיסים מתאים כך שקצוות המולקולה יהיו בהטייה הנכונה להיקשר זה לזה. האורך האופטימלי עבור מולקולת דנ"א מעגלית הוא כ-400 זוגות בסיסיים (136 ננומטר) ומספר שלם של סיבובים, כלומר כפולות שלמים של 10.4 זוגות בסיסים לסיבוב. מולקולה המכילה מספר לא-שלם של סיבובים נתקלת במחסום אנרגיה משמעותי בשביל להפוך למעגלית. למשל, מולקולה המורכבת מ-30 סיבובים כאשר בכל סיבוב יש 10.4 זוגות בסיסים תהפוך למעגלית פי מאות פעמים מהר יותר מאשר זו המכילה 30.5 סיבובים.[37]
מתיחה
משטר מתיחה גמיש
מבחינה אנטרופית, קטעי דנ"א ארוכים גמישים יותר תחת מתח. כאשר מולקולת דנ"א נמצאת בתמיסה, היא עוברת שינויים מבניים מתמשכים באמצעות האנרגיה הזמינה באמבט התרמי של הממס, אודות לרטט התרמי של המולקולה בשילוב עם התנגשויות חוזרות ונשנות עם מולקולות מים. מסיבות אנטרופיות, ככל שהמולקולה הדחוסה רפויה יותר היא זמינה יותר מבחינה תרמית מאשר מולקולה במצב מתוח, ולכן מולקולות דנ"א נמצאות באופן כמעט בלעדי בפריסות רפויות סבוכות.
ניתן למתוח מולקולה אחת של דנ"א תוך השקעת כוח ועל ידי כך ליישר אותה. התנהגות המתיחה של דנ"א נחקרה ונותחה מנקודת מבט של פיזיקה פולימרית באמצעות מלקחיים אופטיים, ופעם נוספת נמצא כי הדנ"א תואם במידה רבה את מודל ה-WLC תחת סולמות אנרגיה הנגישים מבחינה פיזיולוגית.
מעברי פאזה תחת מתיחה
תחת מתח מבוקר ועם מומנט חיובי, משערים כי הדנ"א מקיים מעברי פאזה עם הבסיסים הנוטים כלפי חוץ והפוספטים הנעים למרכז. המבנה המוצע לדנ"א הנמתח יתר על המידה נקרא P-Form DNA, בהוקרה ללינוס פאולינג שהציג את המודל כהשערה למבנה הנפוץ של הדנ"א.[26]
ראיות למתיחה המכנית של דנ"א בהיעדר מומנט מושרה מצביעות על מעבר או מעברים המובילים למבנים נוספים שמוכרים כצורות S-Form DNA. מבנים אלה עדיין לא אופיינו באופן סופי בשל הקושי לבצע הדמיה ברזולוציה אטומית בתמיסות תוך השריית כוח, אך בוצעו מחקרי סימולציה ממוחשבים רבים (לדוגמה,[38],[39]).
המבנים המוצעים של צורת S כוללים מבנים ששומרים על קשרי המערום וקשרי המימן בין זוגות בסיסים (עשירים ב-C ו-G), וכן מבנים בהם מתרחשת דנטורציה חלקית של קשרי המערום אך גאומטריית הבסיסים נשמרת (עשירים ב-A ו-T).
התפתלות־על וטופולוגיה
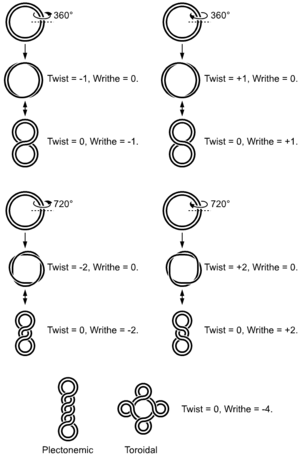
צורה B של סליל הדנ"א משלימה סיבוב של 360° כל 10.4-10.5 זוגות בסיסים (bp) מבלי לגרום למתיחת הסיבוב, אך תהליכים ביולוגיים מולקולריים רבים יכולים לגרום ללחץ מתיחה סיבובי. מקטע דנ"א עם פיתול עודף נקרא "התפתלות־על" (Supercoiled) חיובית ומקטע דנ"א עם פיתול פחוּת נקרא התפתלות־על שלילית. דנ"א in-vivo הוא לרוב בעל התפתלות על שלילית, וניתן לפרום את הסליל הכפול שלו בעזרת רנ"א תעתוק.
בתוך התא רוב הדנ"א מוגבל מבחינה טופולוגית. דנ"א נמצא בדרך כלל בלולאות סגורות (כגון פלסמידים בפרוקריוטים), שנחשבות לסגורות מבחינה טופולוגית, או כמולקולות ארוכות מאוד שמקדמי הדיפוזיה שלהן מייצרים באפקטיביות מתחמים (Domains) סגורים טופולוגית. מקטעים ליניאריים של דנ"א הרבה פעמים נקשרים לחלבונים או למבנים פיזיקליים (כגון ממברנות) על מנת ליצור לולאות טופולוגיות.
פרנסיס קריק היה מהראשונים להציע את חשיבותם של מספרים מקשרים לתיאור התפתולת־על של דנ"א.
ניתוח טופולוגיית הדנ"א מתבסס על שלושה ערכים
- L = מספר המקשר (Linking Number) - מספר הפעמים שגדיל דנ"א אחד עוטף את השני. זהו מספר שלם עבור לולאה סגורה או קבוע עבור תחום טופולוגי סגור.
- T = פיתול (Twist) - סך הסיבובים בסליל הכפול של דנ"א דו-גדילי. לרוב מספר זה יטה לכיוון מספר הסיבובים שסליל דנ"א דו-גדילי יבצע בתמיסה (מספר הבסיסים חלקי 10.5), בהנחה שאין גורמים הנקשרים לחומצות הגרעין (כדוגמת אתידיום ברומיד), או גורמים אחרים המשפיעים על נוקשות הדנ"א.
- W = עיוות (Writhe) - מספר הסיבובים שהסליל הכפול של דנ"א דו גדילי מסתובב סביב ציר סליל העל.
- L = T + W
- Δ L = Δ T + Δ W
כל שינוי של T במתחם טופולוגי סגור חייב להתאזן על ידי שינוי ב-W, ולהפך. התוצאה היא מבנה דנ"א מסדר גבוה יותר. מולקולת דנ"א עגולה (Circular DNA) עם עיוות 0 תהיה מעגלית. אם לאחר מכן נגדיל או נקטין את הפיתול של המולקולה הזאת על ידי התפתלות על, אזי העיוות ישתנה בהתאם.
כאשר שני קצוות של מקטע דנ"א דו גדילי סלילי מחוברים יחד ליצירת מעגל, הגדילים נחשבים לקשורים טופולוגית. משמעות הדבר היא שלא ניתן להפריד בין הגדילים מבלי להביא לפרימת אחד הגדילים לפחות (למשל, באמצעות חום). משימת פרימת הקשירה הטופולוגית של גדילים מחוברים יכולה להתבצע על ידי אנזימים כמו טופואיזומראז. מטרתם של אנזימים אלה לפרום גדיל אחד לפחות מהמעגל כך שמקטע נוסף (חד או דו גדילי) יוכל לעבור דרך המעגל. פרימה זו נחוצה על מנת לאפשר הכפלה של דנ"א מעגלי וכן סוגים שונים של שחלוף בדנ"א ליניארי בעל אילוצים טופולוגיים דומים.
פרדוקס המספר המקשר
במשך שנים רבות, מקורה של התפתלות־העל בגנום של תאים אאוקריוטים היה מעורפל. חידה טופולוגית זו מכונה על ידי כמה "פרדוקס המספר המקשר" (The Linking Number Paradox).[40] עם זאת, כאשר מבנים שנקבעו בצורה ניסויית של הנוקלאוזום הציגו מודל דנ"א מפותל מאוד העוטף משמאל אוקטמר היסטוני,[41][42] הקהילה המדעית החשיבה פרדוקס זה כפתור.
ראו גם
הערות שוליים
- ↑ Kabai, Sándor (2007). "Double Helix". The Wolfram Demonstrations Project.
- 1 2 Alberts (1994). The Molecular Biology of the Cell. New York: Garland Science. ISBN 978-0-8153-4105-5.
- ↑ Wang JC (1979). "Helical repeat of DNA in solution". PNAS 76 (1): 200–203. Bibcode:1979PNAS...76..200W. PMC 382905. PMID 284332. doi:10.1073/pnas.76.1.200.
- 1 2 "Protein-DNA recognition". Annu Rev Biochem 53: 293–321. 1984. PMID 6236744. doi:10.1146/annurev.bi.53.070184.001453.
- ↑ James Watson and Francis Crick (1953). "A structure for deoxyribose nucleic acid". Nature 171 (4356): 737–738. Bibcode:1953Natur.171..737W. PMID 13054692. doi:10.1038/171737a0.(הקישור אינו פעיל, 23.3.2019)
- ↑ "The Complementary Structure of Deoxyribonucleic Acid". Proceedings of the Royal Society of London. 223, Series A: 80–96. 1954.
- ↑ "Secret of Photo 51". Nova. PBS.
- ↑ (הקישור אינו פעיל, 23.3.2019)http://www.nature.com/nature/dna50/franklingosling.pdf
- ↑ The Structure of the DNA Molecule (הקישור אינו פעיל, 23.3.2019)Archived 2012-06-21 at the Wayback Machine
- ↑ "Molecular Structure of Deoxypentose Nucleic (הקישור אינו פעיל, 23.3.2019)[[קטגוריה:דפים עם קישורים שבורים]]Acids|journal=Nature|issue=4356|doi=10.1038/171738a0|year=1953|volume=171|pages=738–740|format=PDF|bibcode=1953Natur.171..738W|pmid=13054693". Wikilink embedded in URL title (עזרה)
- ↑ "On the deoxyribonucleic acid content of sea urchin gametes". Experientia 8 (4): 143–145. 1952. PMID 14945441. doi:10.1007/BF02170221.
- ↑ "Composition of the deoxypentose nucleic acids of four genera of sea-urchin". J Biol Chem 195 (1): 155–160. 1952. PMID 14938364.
- ↑ "The composition of the deoxyribonucleic acid of salmon sperm". J Biol Chem 192 (1): 223–230. 1951. PMID 14917668.
- ↑ Chargaff E (1951). "Some recent studies on the composition and structure of nucleic acids". J Cell Physiol Suppl 38 (Suppl).
- ↑ "The separation and estimation of ribonucleotides in minute quantities". J Biol Chem 186 (1): 37–50. 1950. PMID 14778802.
- ↑ Chargaff E (1950). "Chemical specificity of nucleic acids and mechanism of their enzymatic degradation". Experientia 6 (6): 201–209. PMID 15421335. doi:10.1007/BF02173653.
- ↑ "A proposed structure for the nucleic acids". Proc Natl Acad Sci U S A 39 (2): 84–97. פברואר 1953. Bibcode:1953PNAS...39...84P. PMC 1063734. PMID 16578429. doi:10.1073/pnas.39.2.84.
- ↑ "Nobel Prize - List of All Nobel Laureates".
- ↑ לנינג'ר: עקרונות הביוכימיה, האוניברסיטה הפתוחה, 2018, פרק פרק 8
- ↑ Dickerson RE (1989). "Definitions and nomenclature of nucleic acid structure components". Nucleic Acids Res 17 (5): 1797–1803. PMC 317523. PMID 2928107. doi:10.1093/nar/17.5.1797.
- ↑ "Resolving the discrepancies among nucleic acid conformational analyses". J Mol Biol 285 (4): 1563–1575. 1999. PMID 9917397. doi:10.1006/jmbi.1998.2390.
- ↑ "A standard reference frame for the description of nucleic acid base-pair geometry". J Mol Biol 313 (1): 229–237. 2001. PMID 11601858. doi:10.1006/jmbi.2001.4987.
- ↑ Richmond; Davey, CA (2003). "The structure of DNA in the nucleosome core". Nature 423 (6936): 145–150. Bibcode:2003Natur.423..145R. PMID 12736678. doi:10.1038/nature01595.
- ↑ "The extended and eccentric E-DNA structure induced by cytosine methylation or bromination". Nature Structural Biology 7 (9): 758–761. 2000. PMID 10966645. doi:10.1038/78985.
- ↑ "Application of L-DNA as a molecular tag". Nucleic Acids Symp Ser (Oxf) 49 (49): 261–262. 2005. PMID 17150733. doi:10.1093/nass/49.1.261.
- 1 2 "Stretched and overwound DNA forms a Pauling-like structure with exposed bases". PNAS 95 (24): 14152–14157. 1998. Bibcode:1998PNAS...9514152A. PMC 24342. PMID 9826669. doi:10.1073/pnas.95.24.14152.
- ↑ (הקישור אינו פעיל, 23.3.2019)List of 55 fiber structures Archived 2007-05-26 at the Wayback Machine
- ↑ Bansal M (2003). "DNA structure: Revisiting the Watson-Crick double helix". Current Science 85 (11): 1556–1563.
- ↑ "A glossary of DNA structures from A to Z". Acta Crystallogr D 59 (4): 620–626. 2003. PMID 12657780. doi:10.1107/S0907444903003251.
- ↑ "The chemistry and biology of left-handed Z-DNA". Annual Review of Biochemistry 53: 791–846. 1984. PMID 6383204. doi:10.1146/annurev.bi.53.070184.004043.
- ↑ Sinden, Richard R (15 בינואר 1994). DNA structure and function (מהדורה ראשונה). Academic Press. עמ' 398. ISBN 0-12-645750-6.
- ↑ Ho PS (27 בספטמבר 1994). "The non-B-DNA structure of d(CA/TG)n does not differ from that of Z-DNA". Proc Natl Acad Sci USA 91 (20): 9549–9553. Bibcode:1994PNAS...91.9549H. PMC 44850. PMID 7937803. doi:10.1073/pnas.91.20.9549.
- ↑ "Crystal structure analysis of a complete turn of B-DNA". Nature 287 (5784): 755–8. 1980. Bibcode:1980Natur.287..755W. PMID 7432492. doi:10.1038/287755a0.
- ↑ Stokes, T. D. (1982). "The double helix and the warped zipper—an exemplary tale". Social Studies of Science 12 (2): 207–240. doi:10.1177/030631282012002002.
- ↑ Gautham, N. (25 במאי 2004). "Response to 'Variety in DNA secondary structure'". Current Science 86 (10): 1352–1353. בדיקה אחרונה ב-25 במאי 2012.
However, the discovery of topoisomerases took "the sting" out of the topological objection to the plectonaemic double helix. The more recent solution of the single crystal X-ray structure of the nucleosome core particle showed nearly 150 base pairs of the DNA (i.e., about 15 complete turns), with a structure that is in all essential respects the same as the Watson–Crick model. This dealt a death blow to the idea that other forms of DNA, particularly double helical DNA, exist as anything other than local or transient structures.
(הקישור אינו פעיל, February 2018) - ↑ "Stacked–Unstacked Equilibrium at the Nick Site of DNA". J Mol Biol 342 (3): 775–785. 2004. PMID 15342236. doi:10.1016/j.jmb.2004.07.075.
- ↑ "DNA Dynamics: Bubble ‘n’ Flip for DNA Cyclisation?". Current Biology 15: R377–R379. doi:10.1016/j.cub.2005.05.007.
- ↑ "Molecular dynamics simulation of DNA stretching is consistent with the tension observed for extension and strand separation and predicts a novel ladder structure.". Journal of the American Chemical Society 118: 10989–10994. 1996. doi:10.1021/ja961751x.
- ↑ "Structural basis of pathway-dependent force profiles in stretched DNA.". Journal of Physical Chemistry B 113: 15364–15371. 2009. PMID 19845321. doi:10.1021/jp906749j.
- ↑ Prunell A (1998). "A topological approach to nucleosome structure and dynamics: the linking number paradox and other issues". Biophys J 74 (5): 2531–2544. Bibcode:1998BpJ....74.2531P. PMC 1299595. PMID 9591679. doi:10.1016/S0006-3495(98)77961-5.
- ↑ "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature 389 (6648): 251–260. 1997. Bibcode:1997Natur.389..251L. PMID 9305837. doi:10.1038/38444.
- ↑ "Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 Å resolution". Journal of Molecular Biology 319 (5): 1097–1113. 2002. PMID 12079350. doi:10.1016/S0022-2836(02)00386-8.

רישיון cc-by-sa 3.0
